在砖使用印度生物技术部转换工作
您可以运行你的印度生物技术部核心项目作为一个砖的工作任务。通过运行你的印度生物技术部核心项目的工作任务,可以受益于以下砖的工作特点:
自动化你的印度生物技术部任务和进度工作流,包括印度生物技术部工作。
监控你的印度生物技术部转换并发送通知的状态转换。
包括你的印度生物技术部项目与其他任务工作流。例如,您的工作流可以摄取数据与自动加载程序,与印度生物技术部转换数据,分析数据和一个笔记本电脑的任务。
工件的自动归档工作运行,包括日志,结果,体现和配置。
了解更BOB低频彩多关于印度生物技术部核心,看到印度生物技术部的文档。
开发和生产工作流程
砖建议开发你的印度生物技术部项目砖SQL的仓库。使用砖SQL的仓库,可以测试生成的SQL印度生物技术部和仓库使用SQL查询历史调试查询由印度生物技术部。学习如何使用印度生物技术部核心和dbt-databricks包来创建和运行印度生物技术部项目在您的开发环境中,明白了连接到印度生物技术部核心。
在生产运行您的印度生物技术部的转换,在砖砖推荐使用印度生物技术部的任务工作。默认情况下,印度生物技术部任务将印度生物技术部Python运行过程在一个集群节点工作,和印度生物技术部对选择生成的SQL SQL仓库。
您可以运行印度生物技术部转换serverless SQL仓库或支持SQL仓库,一个通用的集群,或任何其他dbt-supported仓库。本文讨论了前两个选项的例子。
请注意
开发印度生物技术部模型对SQL仓库和生产运行在一个通用的集群性能可能导致细微的差别和SQL语言支持。砖建议使用相同的砖集群运行时版本通用和SQL的仓库。
需求
在砖使用印度生物技术部项目工作,必须设置Git与砖回购的集成。你不能运行一个从DBFS印度生物技术部项目。
你必须有serverless或支持SQL仓库启用。
你必须有砖的SQL权利。
砖建议dbt-databricks包,而不是dbt-spark包。dbt-databricks包是一个叉的砖dbt-spark优化。
创建并运行您的第一个印度生物技术部工作
下面的例子使用了jaffle_shop项目,项目一个例子演示了印度生物技术部核心概念。创建一个工作运行jaffle店项目,执行以下步骤。
去你的砖的着陆页,做以下之一:
点击
 工作流在侧边栏,然后单击
工作流在侧边栏,然后单击 。
。在侧边栏中,单击
 新并选择工作。
新并选择工作。
在任务出现在对话框任务选项卡中,取代添加一个名称为你的工作…对你的工作名称。
在任务名称为任务,输入一个名称。
在类型,选择印度生物技术部任务类型。
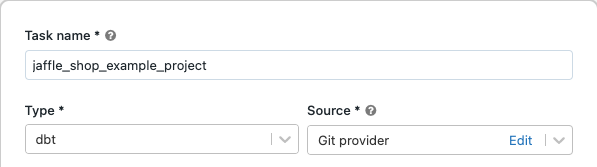
在源,点击编辑并输入jaffle商店GitHub库的详细信息。
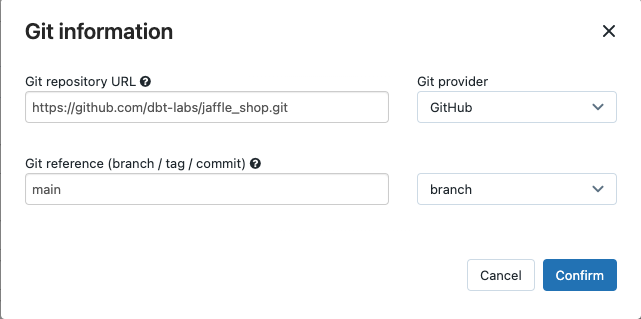
在Git存储库URL,输入的URL jaffle店项目。
在Git参考(分支/标记/提交),输入
主要。您还可以使用一个标签或沙。
点击确认。
在印度生物技术部的命令文本框中,指定印度生物技术部命令来运行(deps,种子,运行)。你必须每个命令前缀
印度生物技术部。命令运行在指定的顺序。
在SQL仓库,选择一个SQL仓库运行生成的SQL印度生物技术部。的SQL仓库下拉菜单显示只有serverless和pro SQL仓库。
(可选)您可以指定一个模式任务的输出。默认情况下,模式
默认的使用。(可选)如果你想改变集群,印度生物技术部核心运行时,点击印度生物技术部CLI集群。集群来最小化成本,默认为一个小型的单节点集群。
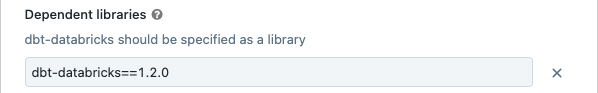
(可选)可以指定dbt-databricks版本的任务。例如,销你的印度生物技术部开发和生产任务到一个特定的版本:
下依赖库,点击
 当前dbt-databricks旁边的版本。
当前dbt-databricks旁边的版本。点击添加。
在添加依赖库,单击PyPI在选项卡并输入dbt-package版本包文本框(例如,
dbt-databricks = = 1.2.0)。点击添加。
请注意
砖建议把你的印度生物技术部任务到一个特定版本的dbt-databricks包以确保相同的版本是用于开发和生产运行。砖建议dbt-databricks的1.2.0或更高版本的包。
点击创建。
现在运行工作,点击
 。
。
印度生物技术部的工作任务的查看结果
当工作完成后,您可以通过运行SQL查询的测试结果笔记本或通过运行查询在你的砖仓库。例如,参见下面的示例查询:
显示表在<模式>;
选择*从<模式>。客户限制10;
取代<模式>任务配置中配置的模式名。
API的例子
您还可以使用乔布斯API创建和管理工作,包括印度生物技术部工作。下面的示例创建一个单一的印度生物技术部的工作任务:
{“名称”:“jaffle_shop印度生物技术部工作”,“max_concurrent_runs”:1,“git_source”:{“git_url”:“https://github.com/dbt-labs/jaffle_shop”,“git_provider”:“gitHub”,“git_branch”:“主要”},“job_clusters”:({“job_cluster_key”:“dbt_CLI”,“new_cluster”:{“spark_version”:“10.4.x-photon-scala2.12”,“node_type_id”:“i3.xlarge”,“num_workers”:0,“spark_conf”:{“spark.master”:“地方(* 4)”,“spark.databricks.cluster.profile”:“singleNode”},“custom_tags”:{“ResourceClass”:“SingleNode”}}}),“任务”:({“task_key”:“转换”,“job_cluster_key”:“dbt_CLI”,“dbt_task”:{“命令”:(“印度生物技术部deps”,“印度生物技术部种子”,“印度生物技术部运行”),“warehouse_id”:“1 a234b567c8de912”},“库”:({“pypi”:{“包”:“dbt-databricks > = 1.0.0, < 2.0.0”}}]}]}
(高级)印度生物技术部运行定制的概要文件
与SQL运行你的印度生物技术部任务仓库(推荐)或一个通用的集群中,使用一个定制的profiles.yml定义仓库或集群连接。创建一个工作运行jaffle店项目仓库或一个通用的集群中,执行以下步骤。
请注意
只可以使用SQL仓库或一个通用集群作为印度生物技术部的目标任务。你不能使用集群工作作为印度生物技术部的一个目标。
创建一个叉的jaffle_shop存储库。
克隆到桌面的分叉的存储库。例如,您可以运行一个命令如下:
git克隆https://github.com/ <用户名> / jaffle_shop.git
取代
<用户名>GitHub处理。创建一个新文件
profiles.yml在jaffle_shop目录包含以下内容:jaffle_shop:目标:databricks_job输出:databricks_job:类型:砖方法:http模式:“<模式>”主机:“< http_host >”http_path:“< http_path >”令牌:”{{env_var (“DBT_ACCESS_TOKEN”)}}”
取代
<模式>与项目的模式名称表。与SQL运行你的印度生物技术部任务仓库,替换
< http_host >与服务器主机名价值的连接细节为你的SQL选项卡仓库。印度生物技术部任务运行通用集群,替换< http_host >与服务器主机名价值的高级选项,JDBC / ODBC集群选项卡为你的砖。与SQL运行你的印度生物技术部任务仓库,替换
< http_path >与HTTP路径价值的连接细节为你的SQL选项卡仓库。印度生物技术部任务运行通用集群,替换< http_path >与HTTP路径价值的高级选项,JDBC / ODBC集群选项卡为你的砖。
你没有指定的秘密,比如访问令牌,在文件中,因为你要检查这个文件源代码控制。相反,这个文件使用印度生物技术部在运行时动态地插入凭证模板功能。
请注意
期间生成的证书是有效的,最多30天,完成后将自动撤销。
检查这个文件到Git并把它发送到你的仓库。例如,您可以运行命令如下:
git添加配置文件。yml git commit - m“添加配置文件。yml砖的工作”git推点击
 工作流侧边栏的砖UI。
工作流侧边栏的砖UI。选择并单击印度生物技术部工作任务选项卡。
在源,点击编辑并输入你的叉形jaffle商店GitHub库的细节。
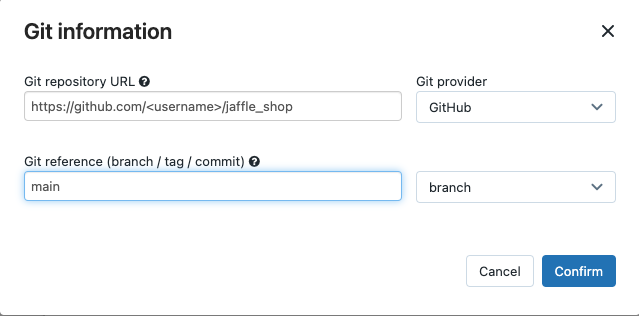
在SQL仓库中,选择没有(手动)。
在配置文件目录,输入目录包含的相对路径
profiles.yml文件。离开路径值留空,使用默认的存储库的根。
(高级)使用印度生物技术部Python在工作流模型
请注意
印度生物技术部支持Python模型是处于测试阶段,需要印度生物技术部1.3或更高版本。
印度生物技术部现在支持Python模型在特定的数据仓库,包括砖。印度生物技术部Python模型,您可以使用Python的生态系统来实现转换的工具很难实现使用SQL。您可以创建一个砖的工作和你的印度生物技术部Python运行一个任务模型,或者你可以包括印度生物技术部任务作为一个工作流,包括多个任务的一部分。
你不能在印度生物技术部任务使用一个SQL运行Python模型仓库。更多信息关于使用印度生物技术部Python模型与砖,明白了特定的数据仓库印度生物技术部的文档。
 。
。



 。
。